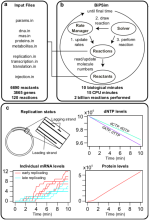
Simuler le comportement stochastique des systèmes biologiques aux échelles les plus fines en un temps de calcul raisonnable est un enjeu majeur en biologie. Face au nombre colossal d’évènements à simuler, des compromis entre granularité des phénomènes simulés, temps de calcul et généricité du simulateur sont nécessaires. Dans ce contexte, les chercheurs du laboratoire MaIAGE de l’INRAE et de l’équipe MAMBA de l’INRIA ont développé le simulateur stochastique open-source BiPSim, combinant une représentation abstraite efficace des réactions et une implémentation en temps constant de l'algorithme de simulation stochastique de Gillespie pour les réactions. BiPSim permet ainsi de simuler efficacement certains processus cellulaires (comme les processus de polymérisation) de façon détaillée et particulièrement délicats à simuler habituellement compte tenu du nombre d’évènements associés. Différents niveaux de granularité dans la description des processus peuvent être traités et simulés simultanément, offrant ainsi un compromis entre temps de calcul et granularité du processus. Conçu pour être générique et donc aisément adaptable à différents types de cellules, BiPSim rend ainsi accessible à la communauté biologique la simulation stochastique de processus cellulaires et permet de confronter les simulations et des données omiques hétérogènes acquises en cellule unique (données de single cell).
Contact : Anne GOELZER et Vincent FROMION - Unité MaIAGE - Centre INRAE IdF Jouy-en-Josas-Antony